II)- Estrutura das proteínas
1)-Estrutura primária das proteínas: A estrutura primária de uma proteína, é a sequência de aminoácidos, unidos por ligação peptídica. Esta estrutura é muito importante, pois muitas doenças genéticas resultam em proteínas com sequências anormais de aminoácidos, ocasionando uma organização irregular, com perda e prejuízo da função normal. Entre nós, o exemplo mais ilustrativo, é a anemia falciforme, uma doença genética, que resulta na produção pelo eritrócito de uma hemoglobina anormal, chamada hemoglobina S. Essa hemoglobina tem as cadeias a normais mas, nas cadeias b, o ácido glutâmico(-) da posição 6, está substituído pela valina (+). Se a estrutura primária das proteínas normais for conhecida, essa informação pode ser usada para diagnosticar ou estudar a doença.
Ligação peptídica:
Nas proteínas, os aminoácidos estão unidos covalentemente por ligações peptídicas, as quais são ligações amida entre o grupo a- carboxila de um aminoácido e o grupo a-amino de outro aminoácido:
A figura representa, a formação da ligação peptídica entre o aminoácido valina e o aminoácido alanina. Como se pode ver, a formação desta ligação covalente, implica a perda de uma molécula de H2O.
Características da ligação peptídica: A ligação peptídia tem um carácter de dupla ligação parcial, isto é mais curta do que uma lig ação simples e é rígida e planar.
Essa configuração rígida planar, impede a rotação livre da ligação entre o carbono carbonila e o nitrogénio da ligação peptídica.
Entretanto, a ligações entre os carbonos a e os grupos a-amino ou a-carboxila podem rodar livremente (embora com certas limitações, provocadas pelo grupo R). Isso permite com a cadeia polipetídica assuma uma variedade de configurações possíveis. A ligação peptídica, é geralmente uma ligação trans, ao invés de cis, em grande parte devido á interferência dos grupos R, uns com os outros, quando em configuração cis.
Polaridade da ligação peptídica: Como todas as ligações amida, os grupos C=O e –NH da ligação peptídica, não possuem carga, nem aceitam ou fornecem prótons na faixa de pH entre 2-12. Assim, os grupos carregados presentes nas proteínas, são simplesmente os seus grupos R e os grupos a-amino terminal e a-carboxilo terminal. Contudo, os grupos C=O e –NH da ligação peptídica, são polares e estão envolvidos em pontes de hidrogénio que estabilizam a estrutura secundária das proteínas.
Nomeando um peptídeo: Por convenção, a extremidade amino livre da cadeia polipeptídica (N-terminal) é escrita á esquerda, e a extremidade carboxila livre (C-terminal), á direita.

Dessa forma, todas as sequências de aminoácidos são lidas da extremidade N-terminal para a extremidade C-terminal. Assim, a direcçaõ de leitura do peptídeo representado abaixo será alanina®arginina e nunca arginina®alanina.
a)-a-Hélice
b)-Folha b
c)-Curvaturasb
d)- Estrutura Secundária não repetitiva
e)- Estruturas supersecundárias.
a)-a-Hélice

Existem várias hélice peptídicas encontradas na natureza, mas a a-hélice é a mais comum. É uma estrutura helicoidal, que consiste em um esqueleto polipeptídico central, em espiral e bem compacto, com as cadeias laterais dos aminoácidos que a compõem, estendendo-se para fora da hélice, de modo a impedir a interferência entre si.
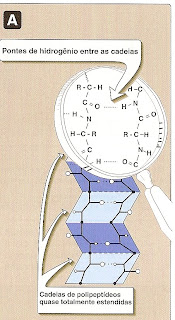
Uma hélice a é estabilizadapor pontes de hidrogénio, entre os átomos de oxigénio dos carbonilas e os átomos de hidrogénio das amidas envolvidas na ligação peptídica, que compõem o esqueleto peptídico, de quatro em quatro resíduos de aminoácidos. Assim cada volta completa (passo) de uma hélice a, contém 3,6 aminoácidos. Existem aminoácidos que quebram a hélice a, como a prolina porque o seu grupo imino não é compatível geométricamente com a espiral. Um grupo de aminoácidos carregados, como o glutamato, aspartato, histidina lisina ou arginina, também quebram a hélice a.
A folha b, é outro tipo de estrutura secundária, na qual todos os componentes da ligação peptídica estão encolvidos com pontes de hidrogénio. As superfícies das folhas b apresentam um aspecto pregueado, motivo pelo qual também são chamadas de folhas b pregueadas.
As folhas b podem ser paralelas (se tiverem o mesmo sentido) ou antiparalelas (de sentido contrário. Quando as pontes de hidrogénio se estabelecem entre duas cadeias polipeptídicas diferentes, elas são chamadas de intercadeias. Quando estas ligações se estabelecem entre aminoácidos da mesma cadeia polipeptídica, são ligações intracadeia.
c)- Curvaturas b (voltas reversas).
As curvaturas b revertem a direcção de uma cadeia polipeptídica, auxiliando a formação de uma estrutura compacta e globular. Elas normalmente são encontradas na superfície das moléculas proteicas e frequentemente contêm resíduos carregados. As curvaturas b geralmente são compostas por quatro aminoácidos, um dos quais pode ser a prolina ou a glicina.
d)- Estrutura secundária não repetitiva
Aproximadamente metade de uma proteína globular média está organizada em estruturas repetitivas como a hélice a ou folhas b. O restante de uma cadeia polipeptídica é decrito como tendo uma conformação em alça ou em espiral.
e)- Estruturas supersecundárias (motivos)
As proteínas globulares são construídas pela combinação de elementos estruturais secundários (hélice a , as folhas b e sequencias não repetitivas). Esses formam principalmente a região central (isso é do interior da molécula). Eles são conectados por regiões em alça (por exemplo curvatura b), na superfície da proteína. As estruturas supersecundárias são normalmente produzidas pelo agrupamento das cadeias laterais estruturais secundários, colocados proximos uns dos outros.
Numa mesma proteína, as as hélice a e as folhas b que estão próximas (adjacentes) uma da outra, devido a sequencia de aminoácidos, também ficarão proximos na proteína após o seu enrolamento final.
3)-Estrutura Terceária da Proteínas
A estrutura primária de uma cadeia polipeptídica, determina a sua estrutura terceária. A estrutura terceária é dada pelo dobramento dos domínios (que são as unidades básicas da estrutura e função das proteínas) e pelo arranjo final dos domínos da cadeia polipeptídica.
As proteínas globulares (cuja estrutura terceária faz lembrar pequenas esferas) quando em solução aquosa, têm um arranjo compacto, com as cadeias laterais hidrofóbicas colocadas no interior do glóbulo, enquanto que os grupos hidrofílicos ficam colocados na superfície. Todos os grupos hidrofílicos (incluindo a ligação peptídica) que ficarem no interior, estarão envolvidos em ligações pontes de hidrogénio ou interações electrostáticas.
a)- Domínios: São as unidades funcionais fundamentais, com estrutura tridimensional em um polipeptídeo. As cadeias polipeptídicas com mais de 200 aminoácidos, têm dois ou mais domínios. O centro do domínio é formado a partir de combinações de elementos estruturais supersecundários (motivos). O dobramento da cadeia peptídica dentro de um domínio, normalmente ocorre independentemente do dobramento de outro domínio.
Interacções que estabilizam a estrutura terceária
A estrutura tridimensional única de cada polipeptídeo é determinada por sua sequência de aminoácidos. As interacções entre as cadeias laterais do aminoácidos direccionam o dobramento do polipeptídeo para formar uma estrutura compacta. Existem quatro tipos de interacções que cooperam para estabilizar as estruturas terceárias das proteínas globulares:
- Pontes dissulfeto. Uma ponte dissulfeto, é uma ligação covalente formada por grupos sulfidrila (-SH) de dois resíduos de cisteína, para formar um resíduo de cistina. As duas cisteínas podem estar separadas uma da outra por vários aminoácidos da mesma cadeia polipeptídica, ou podem mesmo estar situados em cadeias diferentes.
- Interacções hidrofóbicas. Os aminoácidos com cadeias laterais hidrofóbicas, tendem a estar situados no interior da molécula polipeptídica, onde eles se associam com outros aminoácidos hidrofóbicos. Em contraste os aminoácidos com cadeias laterais polares ou com carga, tendem a ficar na superfície da molécula, em contacto com o solvente. Se a proteína estiver localizada num solvente apolar lipídico, o arranjo tende a ser diferente, isto é, os aminoácidos apolares estarão situados no exterior enquanto que os polares estrão situados no interior da molécula. .
- Pontes de hidrogénio. Cadeias laterais de aminoácidos contendo hidrogénio formam pontes de hidrogenio com outras cadeia laterais contendo oxigênio. A formação de pontes de hidrogénio entre grupos polares na superfície de uma proteína e o solvente aquoso aumentam a solubilidade da proteína.
- Interações iónicas. Grupos carregados negativamente como o grupo carboxila (-COO-) na cadeia lateral do aspartato ou do glutamato, podem interagir com grupos carregados positivamente, como grupo amino (-NH3+), na cadeia lateral da lisina.
- Papel das "Chaperoninas" no dobramento proteico
O dobramento final de uma proteína, constituindo a sua estrutura terceária, não depende apenas da sua estrutura primária e das interações que se estabelecem entre os aminoácidos. A prova disso, é que a maior parte das proteínas, depois de desnaturadas, não readquire a sua estrutura terceária original. Isso é devido ao facto de que o dobramento particular de várias proteínas é feito com auxílio de um grupo de proteínas chamadas de chaperoninas. As chaperoninas, são também chamadas de "Heat Shock Proteins" ou proteínas de choque térmico.
Estrutura Quaternária da Proteínas
Muitas proteínas consistem em uma única cadeia polipeptídica, sendo definidas como proteínas monoméricas. Outras, entretanto consistem em duas ou mais cadeias polipeptídicas que podem ser estruturalmente idênticas ou totalmente diferentes. Aassociação dessas unidades polipeptídicas é chamado de estrutura quaternária da proteína. Se existem duas subunidades, a proteína é dimérica, se existem três, é trimérica. Mais do que três é polimérica ou multimérica. As subunidades são mantidas unidas por ligações não covalentes como as pontes de hidrogénio, interações hidrofóbicas, ligações iónicas. As subunidades podem funcionar independentemente umas das outras ou cooperarem umas com as outras, como na hemoglonina. Desnaturação das Proteínas
A desnaturação proteica é o desdobramento e desorganização das estruturas secundária e terceária de uma proteína, mantendo-se intacta a estrutura primária (as ligações peptídicas não se rompem). Os agentes desnaturantes são:
- Calor
- Solventes orgânicos
- Agitação mecânica
- Ácidos ou bases fortes
- Detergentes
- Íons ou metais pesados como o chumbo ou mercúrio.
A desnaturação pode ou não ser reversível. Quando é reversível, a proteína quando colocada novamente em condições ideais, dobra-se novamente em sua estrutura original, assim que agente desnaturante for removido. Entretanto, a maioria das proteínas após a desnaturação não se renatura . As proteínas desnaturadas são insolúveis na água e protanto precipitam em solução.
MÉTODOS DE ESTUDO DAS PROTEÍNAS:
1)-Cromatografia
Método utilizado para separar certas classes de misturas complexas - de compostos orgânicos e inorgânicos – entre uma fase que se move e uma fase estacionária.
É a técnica mais utilizada para separar, identificar e quantificar os aminoácidos presentes em uma mistura, porém pode também ser usada em outros casos, como para separar proteínas com cargas elétricas diferentes, e misturas de carboidratos. Este método leva em consideração as diferenças no sinal e na magnitude das cargas eléctricas líquidas dos aminoácidos em um dado pH, as quais são previsíveis a partir de seus valores de pKa e de suas curvas de titulação.
O método consiste em tomar um longo tubo com partículas de resina sintética insolúvel em água e contendo grupos fixos carregados electricamente – podem ser resinas trocadoras de cátions ou resinas trocadoras de ânions – e colocar por cima desta coluna de resina a solução de aminoácidos em um pH escolhido. O pH influi na afinidade de cada aminoácido pela resina, pois este determina os estado de ionização da molécula. Os aminoácidos num mesmo pH diferem mesmo assim nos valores de pKa dos seus grupos R, o que explica a tendência diferente de cada um para se ligar à resina. Assim, eles se moverão através da coluna em velocidades diferentes. Os aminoácidos que se movem mais rápido – ou seja, ligam-se à coluna com menos força – são eluídos primeiro, e posteriormente os outros, então, uma a uma as frações são coletadas e seus solutos analisados quantitativamente.
Há também a Cromatografia líquida de alto desempenho, que é uma melhoria moderna, com resinas mais fortes e aparelhamento aperfeiçoado que obtém melhores e mais rápidas separações. O processo se dá automaticamente em um aparelho chamado analisador de aminoácidos.
b)-Cromatografia de exclusão por tamanho
Este método - também conhecido como filtração em gel – separa proteínas (ou outras substâncias, como carboidratos) conforme o tamanho das moléculas. A coluna contém esferas de um polímero poroso, sendo assim, quando a mistura proteica é adicionada as moléculas maiores passam mais rapidamente e aparecem nas primeiras frações pois não penetram nos poros das esferas devido ao tamanho; já as menores, penetram pelos poros e demoram mais a migrar.
c)-Cromatografia de afinidade
Esta técnica separa as proteínas por sua especificidades de ligação. A coluna contém um polímero ao qual está unido um ligante específico para a proteína que interessa. Assim, quando se adiciona a mistura de proteínas à coluna as proteínas indesejadas são transportadas pelo líquido eluente, e ficam retidas somente as proteínas que interessam unidas aos ligantes das esferas de polímero. Então esta proteína de interesse é eluída por uma solução contendo o ligante livre.
d)-Cromatografia em Papel
A cromatografia em papel, apesar de haver técnicas mais modernas, é ainda usada na separação de aminoácidos.
Aplica-se a amostra a 5 cm da extremidade da tira de papel de filtro. Essa tira é suspensa em um recipiente hermeticamente fechado, que contém o solvente cromatográfico.
Os solventes para separação de misturas de aminoácidos são misturas de água, álcoois, ácidos ou bases. Quando a migração do solvente chegar até quase ao fim da fita, esta é seca e tratada para permitir a visualização das moléculas de interesse. Há também a cromatografia em papel bidimensional, em que utiliza-se uma folha de papel quadrado ao invés da tira. E o papel é cromatografado em duas misturas de solventes.
2)-Electroforese
Eletroforese é o movimento de solutos carregados eletricamente em resposta à aplicação de um campo elétrico; freqüentemente é usada para separar misturas de íons, proteínas e ácidos nucleicos.
A eletroforese, em adição à cromatografia, é um processo importante para separar proteínas. Neste caso a separação se dá por migração de moléculas em um campo elétrico. Porém, há o incoveniente de geralmente este método inativar as proteínas. As vantagens são que as proteínas são separadas e visualizadas, e permitir a determinação de propriedades cruciais de uma proteína, tais como o seu ponto isoelétrico e seu peso molecular aproximado.
A eletroforese de proteínas é geralmente realizada em géis feitos de polímeros entrecruzados de poliacrilamida, que funciona como uma peneira molecular reduzindo a velocidade de migração das proteínas em proporção aproximada à massa, ou peso molecular, de cada uma delas.
Eletroforese Bidimensional
É a combinação de dois métodos – a eletroforese em SDS (Sódio Dodecil Sulfato) e a focalização isoelétrica – em géis bidimensionais; conseguindo-se assim a resolução de misturas complexas de proteínas.
A focalização isoelétrica é um procedimento usado para determinar o ponto isoelétrico (pI) de uma proteína, estabelece-se um gradiente estável do pH no gel através da aplicação de um campo elétrico, então adiciona-se a solução proteica e o campo elétrico é reaplicado, e assim cada proteína migra até atingir o pH que é igual ao seu pI.
Espectrometria de massa
É um método para identificar os diferentes átomos que compõe uma substância. Um espectrômetro de massa bombardeia uma substância com elétrons para produzir íons, ou átomos eletricamente carregados. Os íons atravessam um campo magnético que curva suas trajetórias de modos diferentes, dependendo de suas massas. O campo separa os íons em um padrão chamado espectro de massa. A massa e a carga dos íons podem ser medidas por sua posição no espectro. Os cientistas identificam assim os elementos e isótopos presentes na amostra. Para as proteínas, esse método serve principalmente para determinar o peso molecularda proteína e assim contribuir para a sua identificação e caracterização.
PCR (Reação em cadeia da polimerase)
Reacção em cadeia da polimerase (em inglês Polymerase Chain Reaction - PCR) é um método de amplificação (de criação de múltiplas cópias) de DNA (ácido desoxirribonucleico) sem o uso de um organismo vivo, por exemplo, Escherichia coli (bactéria) ou leveduras.
O processo de PCR foi descrito por Kary Mullis no final da década de 1980, tendo-lhe sido posteriormente, em 1993, atribuído o Prémio Nobel da Química pelo seu trabalho. Em 1989, a Hoffman La Roche & Perkin-Elmer Corporation patenteou este processo. O método PCR é usado habitualmente nos laboratórios de investigação médica e biológica para uma variedade de tarefas, como a detecção de doenças hereditárias, que é a identificação de "impressões digitais" genéticas, a construção de árvores filogenéticas (árvores de relação entre espécies), a clonagem de genes (ver adiante), testes de paternidade, exames para detecção de agentes patogênicos e etc.
Termociclador, Aparelho utilizado para realizar uma PCR
Aplicações
O PCR encontra sua principal aplicação em situações onde a quantidade de DNA disponível é reduzida. Em teoria, é possível amplificar qualquer DNA. Uma das principais aplicações do PCR é na medicina forense, onde pequenas amostras de DNA retiradas da cena de um crime (pedaços de cabelo, gotas de sangue ou saliva, pedaços de pêlo ou até mesmo a minúscula quantidade de DNA deixada em uma impressão digital) são amplificadas para serem analisadas pelo método de fingerprinting. O PCR também é rotineiramente utilizado em procedimentos científicos de Biologia Molecular como amplificação para gerar mutagênese, detecção de mutações ou preparação de fragmentos de DNA para clonagem (inserção em plasmídeo, por exemplo) como também pode ser utilizado para identificação de patógenos que estão presentes em amostras como por a exemplo identificação de agentes como Cândida sp, Chlamydia trachomatis, HPV Vírus do papiloma humano e seus genótipos, HBV Vírus da Hepatite B. etc O PCR também é utilizado na paleontologia para o sequenciamento genico de animais pré-históricos.
A Reação em Cadeia da Polimerase (PCR) é um método muito sensível de análise e por isso é realizado com muito cuidado para evitar contaminações que possam inviabilizar ou tornar errôneo o resultado. Em primeiro lugar, deve-se extrair o material genético da célula ou outro material a ser estudado (exemplo: vestígios de crimes) sem danificá-lo. Normalmente o material extraído é o DNA (ADN), mas pode-se trabalhar com o RNA (ARN) em uma RT-PCR que é um desdobramento da PCR e possui outras aplicações.
Depois de extraído o DNA, a este é adicionada uma mistura (também conhecida como pré-mix) que contém os dNTPs (desoxirribonucleotídeos trifosfatos), que são as bases nitrogenadas ligadas com três grupos fosfato, os primers também chamados de oligonucleotídeos (ou iniciadores) e a enzima DNA polimerase em uma solução tampão. Toda esta mistura é colocada no termociclador, o qual faz ciclos de temperatura pré-estabelecidos com tempos exatos específicos para cada reação (fragmento a ser amplificado).
Na primeira etapa do ciclo a temperatura é elevada de 94 a 96ºC por pouco tempo para que haja a separação da dupla cadeia de DNA (Desnaturação). Na segunda etapa, a temperatura é reduzida entre 50 a 60ºC dependendo da quantidade de C e G encontrada no primer, para que os primers se anelem (pareiem) com a fita molde de DNA (anelamento). Na última etapa do ciclo a temperatura é elevada a 72ºC para que a enzima possa funcionar sintetizando a nova molécula (extensão), em seguida um novo ciclo é iniciado. Normalmente são realizados de 25 a 40 ciclos para cada reação na qual a taxa de replicação é exponencial 2ciclos
O resultado é analisado através de uma eletroforese em gel de agarose ou de poliacrilamida e depois é interpretado com a ajuda de um profissional competente.
Nested PCR
Utilizado para aumentar a quantidade de produto amplificado final ou para aumentar a sensibilidade da técnica. Suas reações requerem dois pares de primers, um par mais externamente para a 1 reação(round) e outro interno ao produto do 1 round. A reacção de 1 round necessita de um maior tempo de extensao devido ao maior produto a ser amplificado, em seguida adiciona-se uma alíquota do 1 round,que servira de alvo, ao mix de 2 round O produto de 1 round normalmente não é evidenciado em corrida em gel de agarose, por outro lado o produto de 2 round gera grande quantidade de produto e por isso pode ser utilizado na corrida ou para o sequenciamento
A placa contendo o mix e o DNA alvo é "ativado" quando a temperatura atingir 94 graus. Esse procedimento aumenta a especificidade pois a polimerase contem um anticorpo, este anticorpo denatura e ativa a enzima ao atingir a temperatura de 94. Polimerases que nao possuem esse inibidor podem amplificar produtos indesejado(inespecificos)em temperatura ambiente





a sua página é muito interesante. parabens.
ResponderEliminar